
导读
全球生物气候数据的准确性对于理解生物多样性-环境之间关的系是至关重要的。许多研究已经探讨了相关物种分布模型(SDMs)的不确定性及偏差,但是在多种可选择情况下如何有效选择生物气候数据集尚未有研究评估。如何避免由于输入数据的干扰而带来的不切实际的预测,实际上SDMs的准确性是可以通过依据多个数据库生成的生物气候一致性来评估模型预测。针对此,本研究基于多类生物气候数据集探讨了如下问题:(a)首次生成全球尺度生物气候一致性图并分析输入数据一致性对于16种哺乳类物种分布模型可信度的重要性;(b)物种响应曲线(温度和降水)的不一致性(c)由生物气候数据集选择带来的物种分布模型差异。最后(d)研究提供了一种依据生物气候一致性评估模型预测效果的方法。
▉ 原文信息

▉ 正文
气候数据基于卫星影像或者降尺度统计方法要比基于插值方法的成效好,从实地观测来看。基于插值方法的气候数据在气象站很少或不均匀分布的地方会很有问题(例如北极、热带雨林、沙漠及一些山地区域)。卫星影像气候数据也有缺点,一般其预测倾向于更暖和更干。上述说明了物种分布模型选择合适的生物气候数据是一项需要谨慎处理的复杂任务,不要简单地想用一套生物气候数据去应用于所有的区域和尺度。
目前的物种分布模型存在一些不确定性(a)输入数据误差或空缺(物种存在数据)(b)模型特点(算法类型,阈值设置)(c)自然系统动态的内在复杂性(物种迁移能力,种间关系等)。输入数据气候数据的不一致性也是一类重要的不确定性来源会影响生态假设的结果。然而,选择生物气候数据的标准及其影响一直以来都被忽视了,只有少量研究涉及。
基于上述,本研究探讨最新全球气候数据集的生物气候一致性以及生成第一张全球尺度的生物气候一致性图。筛选16种哺乳类以研究不同气候数据集的选择对于物种响应曲线的影响,并计算基于不同气候数据集物种分布范围预测的重叠程度。最后,研究还尝试在建模过程中将生物气候一致性的空间特征纳入,并探讨其对栖息地适宜性预测可靠性的影响。
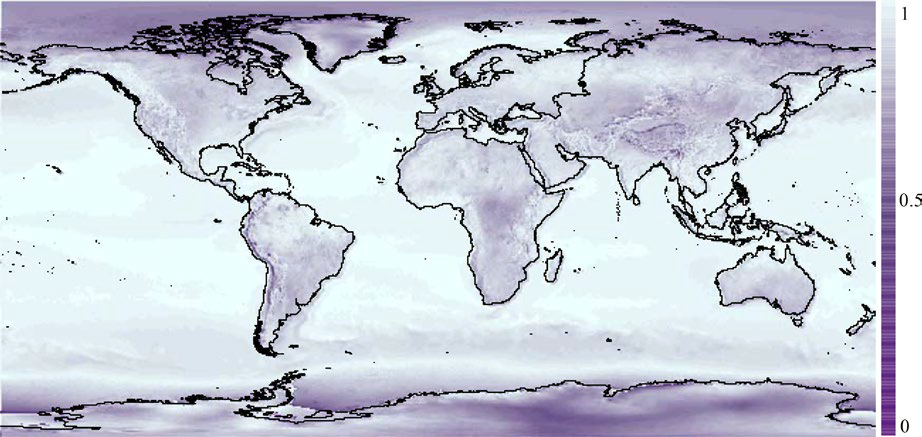
图1 基于Bio- ORACLE v. 2.0, CHELSA, MARSPEC, MERRAclim, WorldClim v. 2数据集生成的全球尺度气候一致性图。
▉ 结果
生物气候一致性图显示在美国西北地区,亚马逊,非洲西北部,古北大陆以及澳洲北部拥有很强的环境一致性;而在极地,安第斯,喜马拉雅,印尼的一些区域以及拉美尼西亚岛则出现生物气候不一致的现象(图1)。
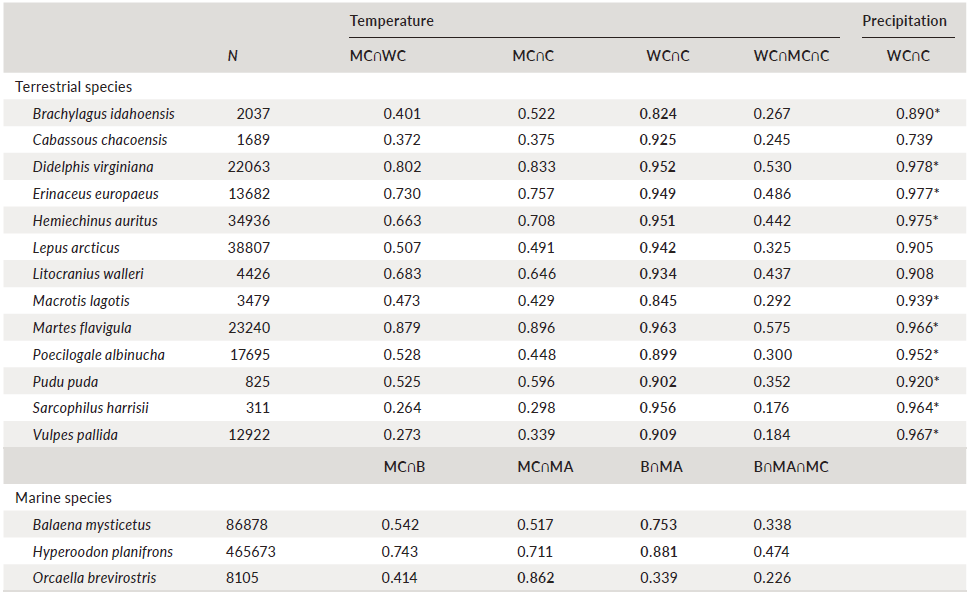
物种对于温度变量的响应在三个数据集(陆地:WorldClim, MERRAclim, 和 CHELSA;海洋:MERRAclim, MARSPEC, and Bio-ORACLE)之间有很大差异,曲线相似性指数(ISC)为0.17-0.58(表1)。陆地哺乳动物中最大的ISC值来源于WorldClim和 CHELSA数据集的温度响应曲线(图2,表2),MERRAclim响应曲线最为特异。而对两种海洋哺乳动物来说,在BioORACLE和MARSPEC数据集的ISC值更大些(图2,表1)。通过比较ISC值可以发现降水响应曲线比温度曲线更为相似(表1,图2,3)。温度物种响应曲线之间较低的ISC值是由于形状和幅度的差异或者二者皆有。
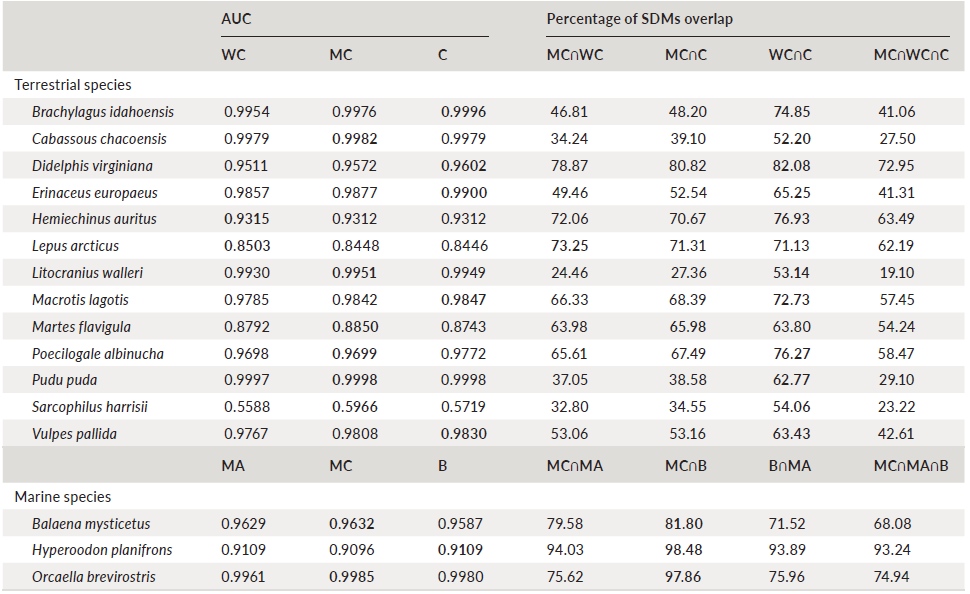
尽管SDM具有很好的模拟效果,但研究还是发现由三类数据集预测的物种分布存在很大差异。对于陆地哺乳动物,分布范围大小(占据的网格单元)与三类数集据预测的物种分布的空间重叠度有显著相关(r=0.76; p<0.01)。也就是,狭域物种预测的分布空间重叠度例较低,而广布物种的空间重叠比例较高(表1,2)。结果还显示WorldClim和CHELSA数据集预测的结果更为相似,在11种陆地哺乳动物中显示出最高比例的空间重叠。而对于海洋哺乳动物则是MERRAclim和Bio- ORACLE数据集(图3)。
L. walleri高度一致性的区域(高适宜性及生物气候一致性)位于索马里、埃塞俄比亚东部、肯尼亚和坦桑尼亚的一些地方(图4c)。然而,一些区域是SDM预测的适宜物种给生存的区域,但却出现很强的气候不一致性。当采用保守的方法筛选90%百分位的生物气候一致性图,三类生物气候数据集预测的物种分布共有的区域约占总面积的65%,提升了前面的空间重叠比例(19%)(表3,图4)。
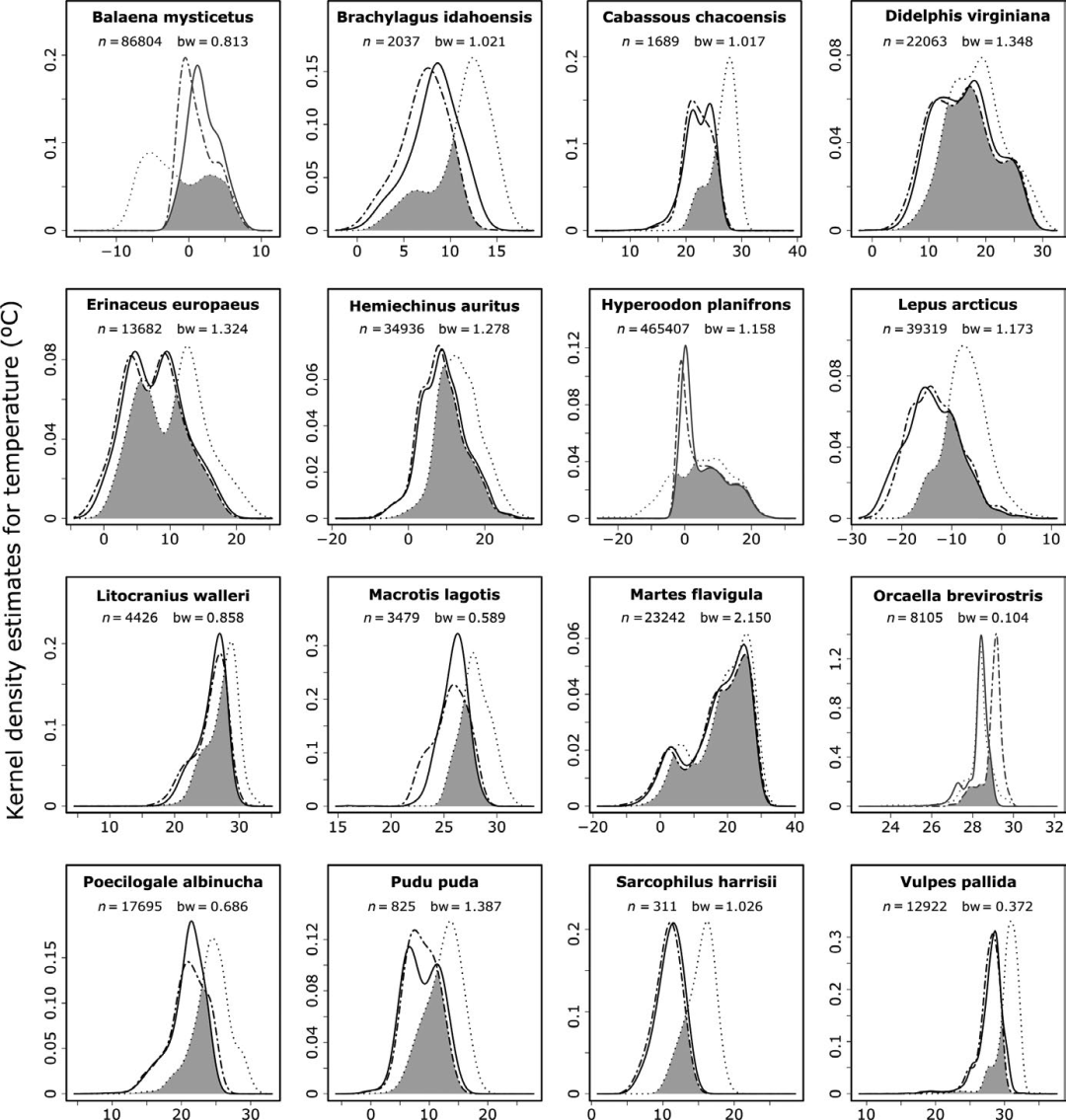
图2 比较基于不同气候数据集获得的物种对年平均温度响应曲线的差异:陆生物种WorldClim(黑色虚线)、CHELSA(黑色实线)及MERRAclim(黑色虚线);以及海洋物种的MERRAclim,MARSPEC(灰色实线)和Bio- ORACLE(灰色虚线)。阴影区域对应于每种情景下选择的三种物种响应曲线(ISC)之间的相似性指数
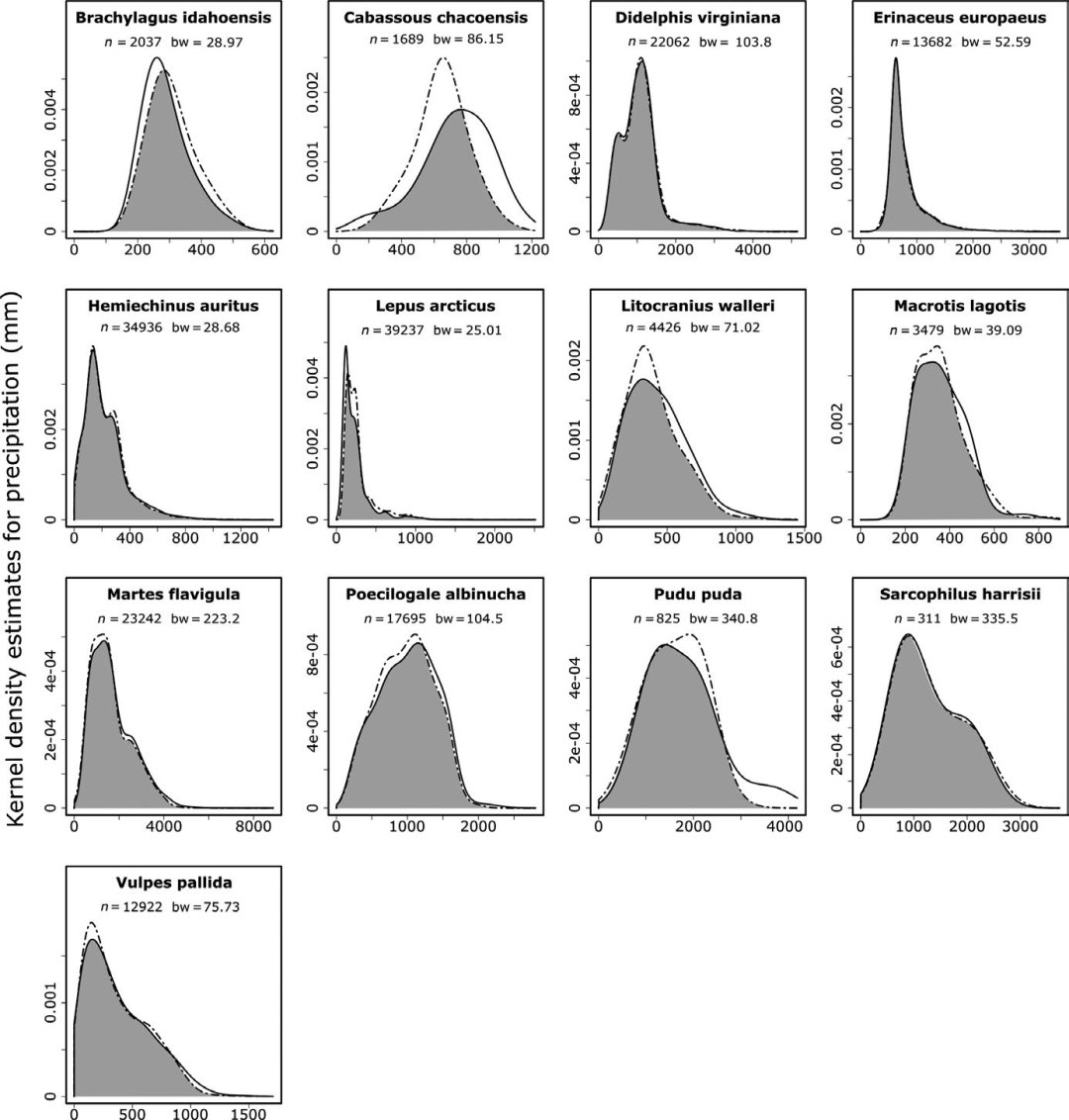
图3 比较基于不同气候数据集获得的物种对年均降水响应曲线的差异。
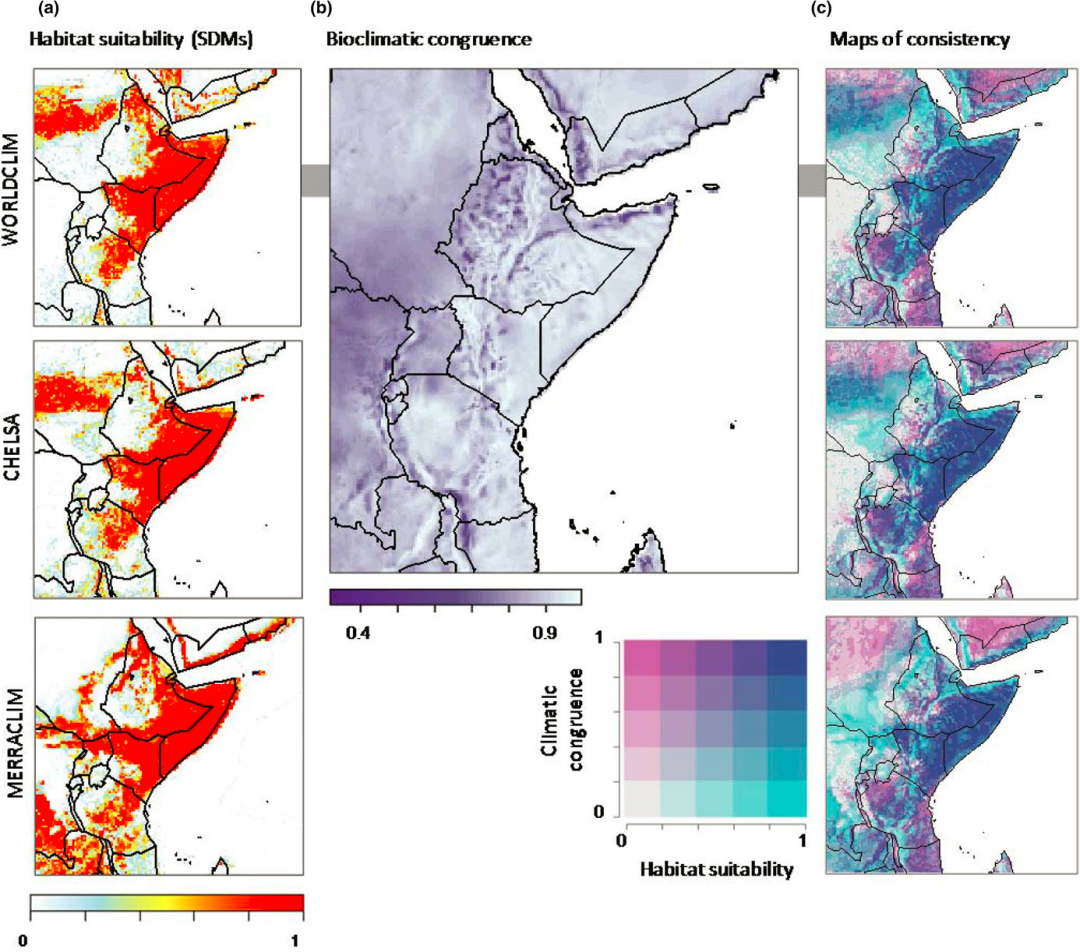
图4 生物气候一致性方法的研究实例:Litocranius walleri。基于WorldClim v. 2,MERRAclim, 和 CHELSA 物种分布模型构建(a)栖息地适宜性图,(b)全球生物气候一致性图,最终得到(c)耦合二者的生物气候学一致性图。
▉ 结论
在分析生物多样性-环境关系时,需要仔细评估和考虑生物气候输入数据的质量。本研究表明,物种模型初始设定条件的微小差异不可避免地导致不同的响应曲线,从而导致对物种空间分布的不可靠预测。这项研究提供了一个经验框架,有效地解释生物多样性分布模型中生物气候数据的局限性,量化其可靠性,并确定采样工作应优先考虑气候不一致区域。这一框架的实施可以澄清许多与物种分布模型相关的问题,并促进它们在宏生态学、生物地理学、古生物学和保护生物学领域的应用。
刘锋 | 云南大学,博士在读
研究方向:气候变化下区域生物多样性保护研究



国务院办公厅关于印发新污染物治理行动方案的通知【国办发〔2022〕15号】









习近平在2020气候雄心峰会上的重要讲话 正式宣布碳排放与新能源装机新目标
